Etiquetas

Las mutaciones se producen en todos los organismos a medida que las células se replican. Los virus ARN mutan más deprisa, cuando el virus hace copias, la enzima que replica el genoma comete errores. La molécula de ADN esta formada por 4 bases: A, G, C y U. La sustitución de los aminoácidos que forman las proteínas superficiales del virus, pueden cambiar las características de propagación y su reacción ante medicamentos antivirales y vacunas.
Los virus de ARN suelen tener tasas más elevadas de mutación que los virus ADN, sin embargo; los coronavirus producen menos mutaciones que la mayoría de los virus ARN porque codifican una enzima que corrige errores cometidos durante la replicación. Incrementa en frecuencia una mutación.
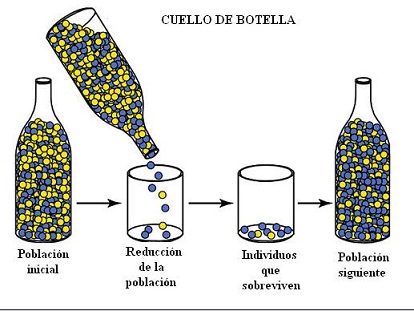
El destino de la mutación esta determinado por la selección natural: si tiene ventaja competitiva respecto a la replicación viral: mejora la transmisión o esquiva la inmunidad; aumentará en frecuencia.
Las mutaciones se suelen producir en la «glicoproteina de pico» que media la unión a las células y es un objeto principal de los anticuerpos neutralizantes. Una mutación puede mejorar la adaptación del virus (unión a receptor) pero también puede perjudicarle si reduce la resistencia a los anticuerpos generados por el huésped.
La genética confirma que aunque los virus viven mutando, su frecuencia de mutación no es muy alta. Es un tipo de virus de ARN que utiliza sus propias enzimas para crear copias de genoma, muta y esa capacidad de mutación es su mayor peligro.

El genoma del virus está asociado con la proteína de la nuclecápside (N).
Esta formado por una cadena de RNA monocatenario de polaridad positiva (+ssRNA) de aproximadamente 30.000 pares de bases.
El genoma se puede dividir en 3 tercios:
- Los dos primeros tercios (próximos al extremo 5´) codifican para el gen de la replicasa viral (constituido por dos ORF (ORF1a y ORF1b), al comienzo de la infección, se traducen en 2 poliproteínas pp1a y pp1ab, procesadas proteolíticamente para generar 16 proteínas no estructurales (nsps) implicadas en la replicación del genomal viral y en la transcripción del RNAm subgenómicos.
- El último tercio del genoma (cercano al extremo 3´) codifica los genes de las 4 proteínas estructurales principales (proteína (S), proteína (M), proteína (E) y proteína (N) y los genes de las proteínas accesorias.

La nueva variante India esta detrás del auge de la pandemia en el sur de Asia(doble mutante del coronavirus la mutación L452R y la mutación E484Q) que le permiten acoplarse con mayor facilidad a las células, como antes fue la británica, subafricana (N501Y) y brasileña. Se ha expandido desde China a los demás continentes.

La teoría de Darwin de la evolución presentada en 1859 requiere variaciones hereditarias de la especie para adaptarse al medio. Se relaciona con las mutaciones del gen, con efectos hereditarios, así con cambios causados en nuevos alelos con fenotipos nuevos podemos identificar el gen. Como las mutaciones son deletéreas, una mutación demasiado frecuente supondría una desventaja para los individuos que la sufriesen. Una tasa de mutación demasiado baja no aporta novedades ventajosas adaptativas para el avance evolutivo en condiciones nuevas, las velocidades de mutación suelen ser óptimas para el organismo.
Todos los organismos tienen aproximadamente la misma frecuencia de mutación por replicación, tanto si son grandes como si son pequeños. La tasa de mutación del genoma como un todo consiste en la suma de las tasas de mutación de todos los genes.
N: número total de genes
U: Tasa de mutación de todos los genes juntos
u: genes aislados con tasa de mutación
N= U/u
Drake ha estimado una tasa de mutación por par de bases mucho mayor para genes particulares de los virus que de las bacterias. No da diferencia significativa si se tiene en cuenta que los números de pares de bases son mayores en las bacterias que en los virus.
CEPA: es cuando los cambios en la virulencia tienen diferentes propiedades biológicas hasta ahora SARS-CoV-2 tiene diferentes secuencias de genomas pero las mismas propiedades biológicas, son la misma cepa.
VARIANTE: cuando los virus tienen varias mutaciones que hacen que se vayan agrupando en ramas filogenéticas.
TASA DE MORTALIDAD: se toma como referencia la población total.
TASA DE LETALIDAD: solo se tiene en cuenta las personas afectadas por la enfermedad.
TASA DE MUTACIÓN: frecuencia de más mutaciones en un solo gen u organismo a lo largo del tiempo.
LA TASA DE MUTACIÓN ALTA: aumenta el número de mutaciones perjudicales o deletereas de los individuos. Cada especie tiene una «tasa de mutación» que ha sido modulada por la «seleccion natural» para enfrentarse a la estabilidad de cambio en el ambiente.
Las tasas de mutación espontáneas son 10 elevado a -5 o 10 elevado a -6 por gen y generación.
Bibliografía:
International Committee on Taxonomy of Viruse: ICTV: «Coronavirus»
https://talk.ictvonline.org/search-124283882/?q=coranovirus#gsc.tab=0&gsc.q=coranovirus&gsc.page=1
Nuno, Rodrigues Faria, “COVID-19 complete virus genomes,” University Oxford, United Kingdom- Univ. Sao Paulo, Brasil, Virological.org 2020
http://virological.org/t/first-cases-of-coronavirus-disease-covid-19-in-brazil-south-america-2-genomes-3rd-march-2020/409
OMS: «Tasa de letalidad COVID-19 «
https://www.who.int/bulletin/volumes/99/1/20-265892-ab/es/
GISAID
https://www.gisaid.org/
Drake, J.W. , “Comparative rates of spontaneous mutation”. Nature., 1969
Strickberger, Monroe W.“Genética” Universidad de Missouri. Ed. Omega, 1978